Recherche
Je suis présentement doctorant en génie électrique et informatique, quoique je me spécialise en biologie computationnelle. Je m'intéresse tout particulièrement aux applications de l'apprentissage automatique dans la conception d'inhibiteurs peptidiques et l'étude de la méthylation des lysines. Dans le cadre de mes projets de recherches, j'utilise des techniques de calcul haute performance (high performance computing). Mes directeurs de thèse sont les professeurs James R. Green (département de génie des systèmes et informatique) et Kyle K. Biggar (département de biochimie).
Recherche actuelle
Dans le cadres de mes travaux de thèse, je m'intéresse aux applications de l'IA pour l'étude de la méthylation des lysines. Mes travaux se déclinent en trois axes de recherche:
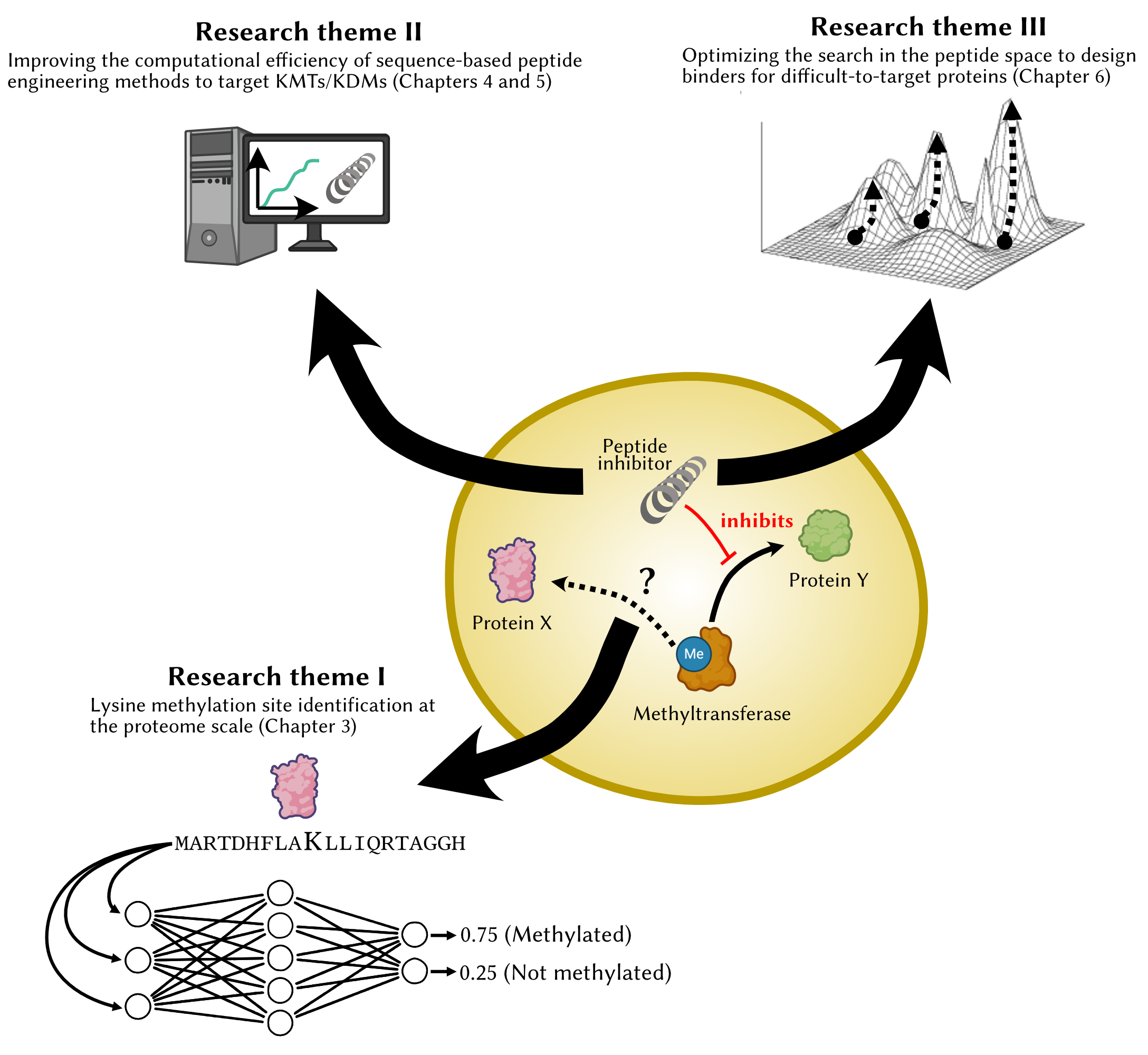
- Identification du méthylome des lysines à l'échelle du protéome avec l'IA
Nos cellules expriment au-delà de 20,000 différentes protéines qui sont sujettes à des modifications chimiques appellées modifications post-traductionnelles (phosphorylation, acétylation, méthylation, etc.) qui altèrent leur fonction, leur stabilité et leur localisation dans la cellule. Quelles protéines sont sujettes à la méthylation sur leurs lysines? Quel est le lien entre la méthylation et la fonction? - Développement d'algorithmes de conception d'inhibiteurs peptidiques
ciblant les (dé)méthylases de lysines plus performants
J'itère sur l'état de l'art en algorithmes de conception d'inhibiteurs peptidiques. Les algorithmes qui sont informés par les séquences en acides aminés qui existent présentement ont des coûts de calculs très élevés. Nous développons un algorithme plus performant nommé Darwin qui fait usage de grandes quantités de données sur les interactions entre les protéines pour concevoir des inhibiteurs de (dé)méthylases des lysines. Ces enzymes sont surexprimées dans un nombre important de cancers et nous anticipons que ces peptides pourraient être utilisés comme thérapies adjuvantes pour augmenter l'efficacité des traitements actuels. - Optimisation de l'algorithme de conception d'inhibiteur peptidiques pour
les protéines difficiles à cibler
Les algorithmes de conception d'inhibiteurs peptidiques qui utilisent la structure primaire des protéines font usage de banques de données répertoriant les interactions entre protéines. Dans certains cas, les protéines ciblées ont peu de similarité avec les protéines ayant des interactions connues, ce qui rend la conception d'inhibiteurs plus difficile. J'essaie donc d'optimiser la stratégie avec laquelle l'espace (infiniment grand) des peptides est exploré pour converger aussi rapidement que possible vers des peptides capables d'interagir avec la cible en utilisant des techniques d'apprentissage profond (modèle de langage des protéines).
Travaux antérieurs
- Structural biology
Sous la supervision du professeur Jean-François Couture, j'ai complété ma thèse de premier cycle sur la caractérisation structurale de la protéine Fur (Ferric uptake regulator) dans C. jejuni. J'ai participé à la résolution de la structure de la protéine. Les résultats des travaux auxquels j'ai contribué ont été publiés dans la revue scientifique Scientific Reports. - Audiologie computationnelle
J'ai complété des travaux en informatique biomédicale dans le cadre de mon projet de maîtrise. Je me suis intéressé à la classification automatique de courbes audiométriques pour faciliter leur interprétation par des professionnels de la santé non-spécialistes.